薬剤耐性・バイオフィルム
薬剤耐性遺伝子の動態解明・拡散抑制手法の開発
本研究の背景と目的
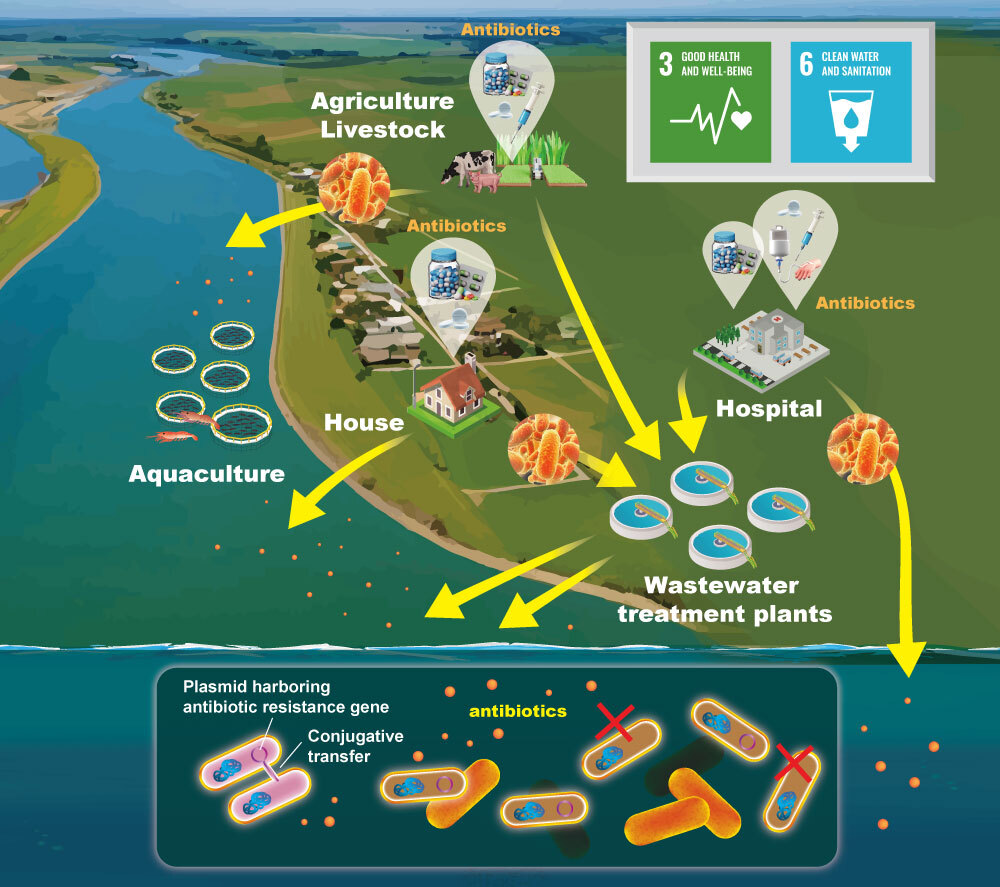
抗生物質の過剰および不適切な使用により、抗生物質が効かない薬剤耐性菌が出現し、その蔓延が世界的な課題となっています。現在、薬剤耐性菌感染症による死者数は年間およそ70万人と推定されており、このままでは2050年には年間1,000万人に達し、現在の癌による死者数を上回る可能性が示唆されています。
薬剤耐性遺伝子は、プラスミドのような移動可能なDNA上に存在することが多く、細菌が外部からDNAを取り込む「水平伝播」という現象によって、さまざまな細菌種へ広がります。本研究では、自然環境中で薬剤耐性遺伝子がどのような細菌種にどの程度伝播しているのかを解明するとともに、薬剤耐性遺伝子の水平伝播を抑制する手法の開発を目指します。
バイオフィルム形成メカニズムの解明
バイオフィルムとは、微生物が自ら生成する細胞外代謝産物によって、固体表面や生体の表面に付着して形成される微生物の集合体であり、様々な場所に存在し、さまざまな問題を引き起こしています。例えば、下水管の腐食や膜を利用した水処理において問題となるバイオファウリングは、このバイオフィルムが原因であると考えられています。これらのバイオフィルムは物質表面に強固に付着し、化学物質に対する耐性を有するため、完全に除去することが難しいとされています。バイオフィルムを完全に除去するためには、微生物がどのようにしてバイオフィルムを形成するか、そのプロセスを理解する必要がありますが、現時点ではそのメカニズムはほとんど解明されていません。また、薬剤耐性菌の多くがバイオフィルムの内部で出現していることも明らかにされつつありますが、いつ、どこで、どのように薬剤耐性菌がバイオフィルム内部で出現しているのか具体的な動態は明らかにされていません。そこで、当研究室では、バイオフィルムの形成メカニズムを遺伝子レベルで解析し、その詳細を明らかにすることを目指しています。
論文情報 (詳細は業績をご覧ください)
Takahashi K, Oshiki M, Ruan C, Morinaga K, Toyofuku M, Nomura N, Johnson DR.
Environ Microbiol Rep. 2023 16:e13221.
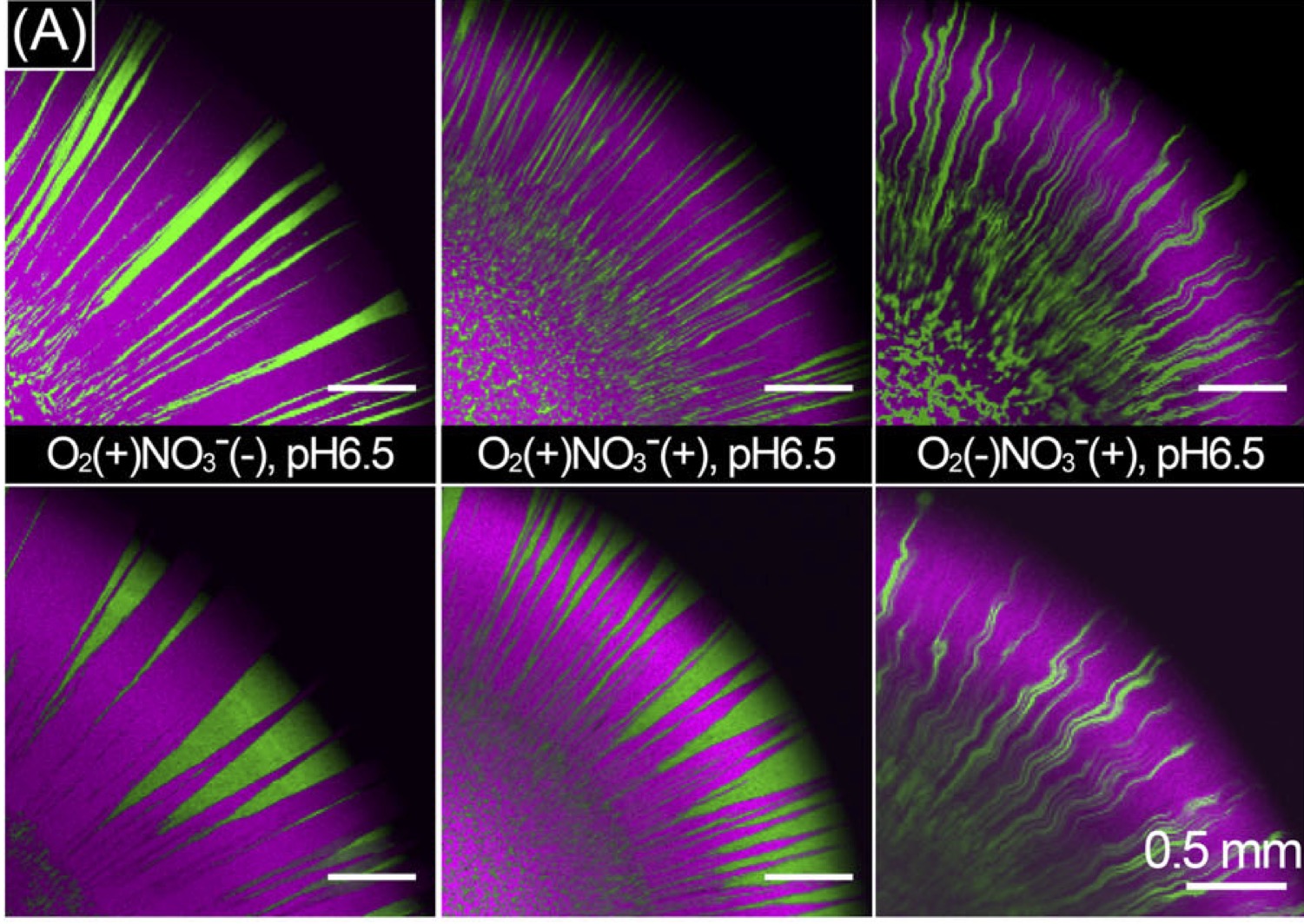
本研究では、低酸素環境における脱窒が窒素酸化物の中間体(亜硝酸や一酸化窒素)の蓄積を増加させ、微生物集団の進化的潜在力に与える影響について調査しました。Paracoccus denitrificansをモデル微生物として実験を行い、実験室内の培養と個別ベースの計算モデルを組み合わせて解析しました。その結果、低酸素環境下では亜硝酸や一酸化窒素の蓄積が増加し、これらの中間体が低pH条件下での細菌の成長に悪影響を与えることが確認されました。 特に、この環境下では細菌がバイオフィルムやコロニーを形成する傾向が見られ、これにより中間体の局所的な蓄積がさらに促進されました。バイオフィルムやコロニー形成によって細菌はより高密度に集まり、これが集団内の細胞間相互作用を活性化し、遺伝的多様性を保持しやすくすることが示されました。結果として、初期集団からの遺伝的多様性が高まり、自然選択の対象となる遺伝的ターゲットが増加し、進化的潜在力が高まることが明らかになりました。 この研究は、低酸素環境における脱窒が微生物のバイオフィルム形成やコロニー形成といった集団行動に影響を与え、窒素酸化物の蓄積が進化的プロセスに与える影響を解明する重要な知見を提供します。これにより、自然および人工環境における窒素循環の制御や微生物集団の管理に有用な情報が得られます。
より詳細な情報は以下のページをご覧下さい
Article page
Oshiki M, Saito T, Nakaya Y, Satoh H, Okabe S.
J Biosci Bioeng. 2023 136:430–437.
本研究では、好気性アンモニア酸化細菌であるNitrosomonas europaeaの細胞がバイオフィルムおよび浮遊状態で増殖した際の、細胞外高分子物質(EPS)の産生とトランスクリプトーム応答について評価しました。N. europaeaは、窒素循環においてアンモニアを亜硝酸に酸化する役割を担い、特に下水処理において生物学的窒素除去の重要なステップとなります。本研究では、無機培地と有機培地(NR培地)を用い、細胞がそれぞれ浮遊およびバイオフィルムの形態で増殖することを確認しました。バイオフィルムモードでの増殖では、EPSの産生量が増加し、EPSの組成は赤外分光法(FT-IR)や核磁気共鳴(NMR)を用いて詳細に解析されました。
RNA-Seq解析により、バイオフィルムモードでは、アンモニアモノオキシゲナーゼ(amo)、ヒドロキシルアミンデヒドロゲナーゼ(hao)、銅含有亜硝酸還元酵素(nirK)など、窒素代謝に関与する遺伝子の転写量が増加していることが示されました。また、細菌の凝集形成に関与するpepAやEPSの移動を助けるwza遺伝子も、バイオフィルムモードで高い転写量が観察されました。これらの結果は、N. europaeaが環境条件に応じてEPS産生を調整し、効率的な窒素酸化活性を維持するための適応戦略を持っていることを示しています。この研究は、N. europaeaのバイオフィルム形成メカニズムを解明し、高密度細胞維持による窒素除去プロセスの効率化に貢献する重要な知見を提供します。

より詳細な情報は以下のページをご覧下さい
Article page
論文等の業績
Key Achievements
- Generation of small colony variants in biofilms by Escherichia coli harboring a conjugative F plasmid
Tashiro, Y., Eida, H., Ishii, S., Futamata, H., Okabe, S.
Microbes Environ., 2017, 32, 40-46. - Increased antibiotic resistance of Escherichia coliin mature biofilms
Ito, A., Taniuchi, A., May, T., Kawata, K., and Okabe, S.
Appl. Environ. Microbiol., 2009, 75(12), 4093-4100. - Induction of multidrug resistance mechanism in Escherichia coli biofilms by interplay between tetracycline and ampicillin resistance genes
May, T., Ito, A., and Okabe, S.
Antimicrob. Agents Chemother., 2009, 53(11), 4628-4639. - Escherichia coli harboring a natural IncF conjugative F-plasmid develops complex mature biofilms by stimulating synthesis of colanic acid and curli
May, T., and Okabe, S.
J. Bacteriol., 2008, 190(22), 7479-7490.


